Amminoacidi e proteine
Da un punto di vista chimico le proteine non sono altro che delle poliammidi
dove le unità monomere sono costituite da ![]() -amminoacidi.
Gli amminoacidi che si ottengono dall'idrolisi acida o basica delle proteine
(una ventina) sono tutti caratterizzati, a parte la glicina che è
il più semplice di essi, da un carbonio chirale al quale sono legati
i due gruppi funzionali NH2 e COOH. Questo carbonio ha sempre
configurazione L. Tale tipo di notazione, come già fatto notare
nel caso degli zuccheri, sta a indicare che il carbonio
-amminoacidi.
Gli amminoacidi che si ottengono dall'idrolisi acida o basica delle proteine
(una ventina) sono tutti caratterizzati, a parte la glicina che è
il più semplice di essi, da un carbonio chirale al quale sono legati
i due gruppi funzionali NH2 e COOH. Questo carbonio ha sempre
configurazione L. Tale tipo di notazione, come già fatto notare
nel caso degli zuccheri, sta a indicare che il carbonio
![]() degli amminoacidi ha la stessa configurazione del carbonio chirale della
L-gliceraldeide. La formula generica di un
degli amminoacidi ha la stessa configurazione del carbonio chirale della
L-gliceraldeide. La formula generica di un ![]() -amminoacido
è mostrata in Figura 11, dove la configurazione del centro chirale
è messa in evidenza tramite una proiezione di Fisher ed è
paragonata a quella della L-gliceraldeide.
-amminoacido
è mostrata in Figura 11, dove la configurazione del centro chirale
è messa in evidenza tramite una proiezione di Fisher ed è
paragonata a quella della L-gliceraldeide.
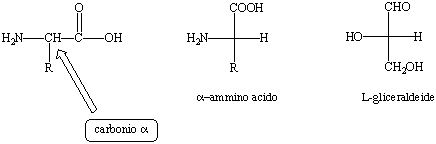
Figura 11. Formula generale di un ![]() -amminoacido.
La configurazione del carbonio chirale (carbonio
-amminoacido.
La configurazione del carbonio chirale (carbonio ![]() )
è messa in evidenza nella proiezione di Fisher e paragonata a quella
della L-gliceraldeide.
)
è messa in evidenza nella proiezione di Fisher e paragonata a quella
della L-gliceraldeide.
Le cellule utilizzano i diversi ![]() -amminoacidi
per sintetizzare le proteine necessarie. Gli amminoacidi, a loro volta,
possono essere sintetizzati da tutti gli organismi viventi. Tuttavia alcune
specie biologiche, fra cui l'uomo, non sono in grado di sintetizzare alcuni
degli amminoacidi che compongono le rispettive catene proteiche. Questi
amminoacidi essenziali devono quindi essere recuperati dall'ambiente
esterno tramite la dieta. Le proteine (animali e vegetali) che vengono
così ingerite, sono idrolizzate e gli amminoacidi ottenuti vengono
ricombinati nell'ordine richiesto per la sintesi delle proteine necessarie.
Nel caso degli umani gli amminoacidi essenziali sono otto: Valina (Val),
Leucina (Leu), Isoleucina (Ile), Fenilalanina (Phe), Triptofano (Trp),
Treonina (Thr), Metionina (Met), Lisina (Lys). La loro struttura è
mostrata in Figura 12. Entro parentesi è riportato il simbolo con
il quale questi amminoacidi vengono comunemente indicati.
-amminoacidi
per sintetizzare le proteine necessarie. Gli amminoacidi, a loro volta,
possono essere sintetizzati da tutti gli organismi viventi. Tuttavia alcune
specie biologiche, fra cui l'uomo, non sono in grado di sintetizzare alcuni
degli amminoacidi che compongono le rispettive catene proteiche. Questi
amminoacidi essenziali devono quindi essere recuperati dall'ambiente
esterno tramite la dieta. Le proteine (animali e vegetali) che vengono
così ingerite, sono idrolizzate e gli amminoacidi ottenuti vengono
ricombinati nell'ordine richiesto per la sintesi delle proteine necessarie.
Nel caso degli umani gli amminoacidi essenziali sono otto: Valina (Val),
Leucina (Leu), Isoleucina (Ile), Fenilalanina (Phe), Triptofano (Trp),
Treonina (Thr), Metionina (Met), Lisina (Lys). La loro struttura è
mostrata in Figura 12. Entro parentesi è riportato il simbolo con
il quale questi amminoacidi vengono comunemente indicati.
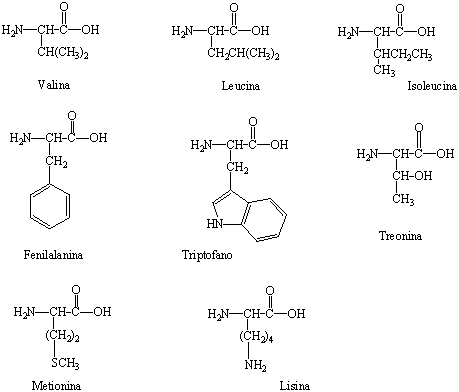
Figura 12. Struttura degli amminoacidi essenziali.
La sequenza con cui i vari amminoacidi si susseguono lungo la catena proteica costituisce la struttura primaria della proteina. Quando le catene polimeriche che si ottengono dall'unione dei vari amminoacidi non sono molto lunghe (peso molecolare inferiore a 10.000) si utilizza il termine polipeptide, anziché quello di proteina. Più precisamente una molecola formata da due amminoacidi legati da un legame ammidico si chiama dipeptide, se gli amminoacidi sono tre (quindi con due legami ammidici presenti) si usa il termine tripeptide, e così via fino a dieci. Per un numero di amminoacidi compreso fra dieci e venti si usa il termine oligopeptide, mentre il termine polipeptide è riservato a molecole formate da molte decine di amminoacidi. La successione di amminoacidi, saldati fra di loro da legami ammidici (comunemente indicati con il termine di legami peptidici) viene precisata utilizzando in sequenza i simboli caratteristici dei vari amminoacidi. Per convenzione i vari amminoacidi vengono scritti a partire da sinistra cominciando dall'amminoacido che ha il gruppo NH2 libero (ovvero non impegnato in un legame peptidico) e procedendo verso destra in direzione dell'amminoacido con il gruppo COOH libero. L'amminoacido a sinistra (NH2 libero) viene indicato come amminoacido N-terminale, mentre quello a destra (gruppo COOH libero) è detto amminoacido C-terminale.
La sequenza Gly-Ser-Ala, ad esempio, indica un tripeptide in cui una molecola di glicina (amminoacido N-terminale) è legata ad una molecola di serina (in posizione centrale) e questa, a sua volta, ad una molecola di alanina (amminoacido C-terminale). I legami peptidici coinvolti sono ovviamente due: Gly-Ser e Ser-Ala. La struttura del tripeptide Gly-Ser-Ala è mostrata in Figura 13, con l'indicazione dei legami peptidici presenti.
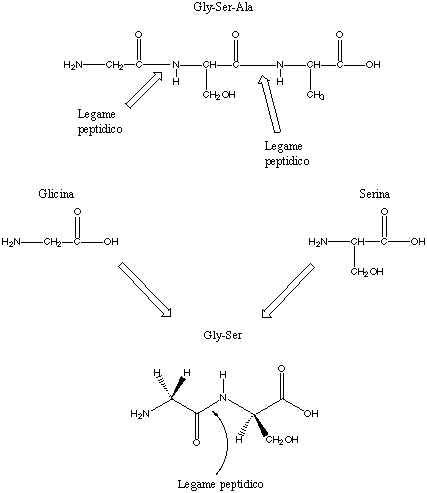
Figura 13. Struttura del tripeptide Gly-Ser-Ala (in alto) e formazione
del legame peptidico fra le due unità di glicina e serina.
È necessario, a questo punto, esaminare in dettaglio la natura del legame peptidico (caratteristiche geometriche ed elettroniche). Una comprensione di questi aspetti è indispensabile per rendersi pienamente conto dei livelli di complessità superiori alla struttura primaria, che vengono generati dal ripiegamento della catena polipeptidica nello spazio (struttura secondaria e terziaria della proteina). A questo proposito consideriamo in dettaglio il legame peptidico di Figura 13 fra le due unità di glicina e serina. Il dimero Gly-Ser è rappresentato in modo da metterne in evidenza le caratteristiche strutturali fondamentali. È evidente come tutti gli atomi legati direttamente al carbonio e all'azoto della funzione ammidica siano sullo stesso piano (piano del foglio). I soli legami che fuoriescono dal piano sono quelli fra i carboni tetraedrici e i sostitutenti (due atomi di idrogeno per la glicina e un atomo di idrogeno ed un gruppo CH2OH per la serina). Queste caratteristiche strutturali sono confermate da studi cristallografici ai raggi X i quali mettono in evidenza altri aspetti interessanti: per esempio il legame C-N è insolitamente corto per un legame singolo (1,32 Å) e il legame C=O (1,23 Å) un po' più lungo di un normale legame carbonilico. Inoltre gli angoli di legame attorno all'atomo di azoto ammidico sono molto vicini a 120° e non a 109° come ci si può aspettare per un azoto che forma tre legami in uno stato di ibridizzazione approssimativamente di tipo sp3. Questa geometria può essere giustificata assumendo che il legame peptidico (e in generale il legame ammidico) possa essere descritto accuratamente solo come un ibrido di risonanza delle due formule limiti I e II mostrate in Figura 14. La formula limite I mette in evidenza un doppio legame carbonio-ossigeno, mentre lo stesso legame è descritto come singolo nella formula limite II. Analogamente il legame fra il carbonio carbonilico e l'atomo di azoto è singolo in I, ma diventa doppio in II. Il movimento del doppietto elettronico dall'azoto alla regione del legame C-N e il conseguente spostamento del doppietto p sull'atomo di ossigeno comporta una separazione di carica in II (azoto positivo e ossigeno negativo). Come è ben noto dalla teoria della risonanza la molecola reale non corrisponde né a I né a II, ma è una combinazione delle caratteristiche di entrambe le formule limiti. Così il legame C-N avrà nella realtà un non trascurabile carattere di doppio legame (il che spiega il legame insolitamente corto) e l'azoto avrà una struttura più o meno planare, con angoli vicini a 120°, necessaria per permettere la coniugazione del doppietto elettronico. L'accentuato carattere di doppio legame presente nel legame peptidico impedisce di fatto una libera rotazione attorno all'asse C-N e mantiene questi due atomi e i quattro atomi ad essi legati tutti sullo stesso piano. È invece libera la rotazione attorno ai legami fra il carbonio carbonilico e il carbonio tetraedrico (chirale) e fra quest'ultimo e l'atomo di azoto. Queste caratteristiche strutturali sono di importanza fondamentale nel determinare la struttura secondaria del polipeptide o della proteina.
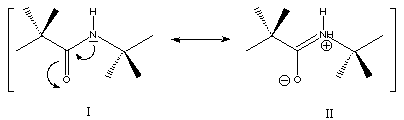
Figura 14. Strutture limiti che descrivono il legame peptidico.
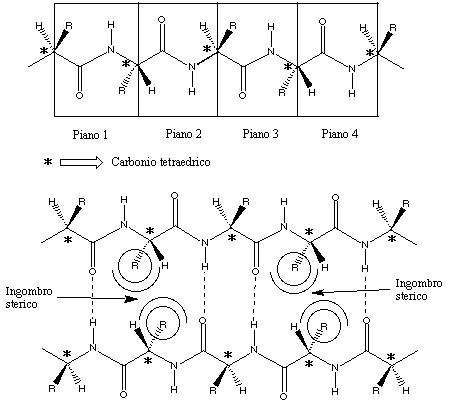
Figura 15. Ipotetica struttura planare di catene polipeptidiche. Nella
parte in basso è messa in evidenza l'interazione di due catene,
o due segmenti della stessa catena, tramite legami idrogeno.
La struttura secondaria riguarda l'orientazione nello spazio
dei vari amminoacidi in regioni localizzate di una catena polipeptidica.
Consideriamo una ipotetica catena di cui rappresentiamo in Figura 15 (in
alto) un breve tratto composto da cinque generici amminoacidi, mettendo
in evidenza per ogni monomero l'orientazione corretta dei vari legami.
Come si vede la struttura complessiva corrisponde ad una successione di
piani (un piano per ogni legame peptidico) tutti coincidenti con il piano
del foglio e separati dai gruppi CHR (carbonio tetraedrico chirale). Poiché
l'ossigeno del carbonile e l'idrogeno del gruppo NH sono in posizione relativa
trans, i gruppi R si trovano alternativamente da una parte e dall'altra
rispetto al piano generale dell'intera struttura. Poiché la catena
si snoda nello spazio con successivi ripiegamenti (una specie di nastro
ripetutamente ripiegato su se stesso), immaginiamo che due segmenti della
stessa catena si dispongano paralleli accoppiandosi tramite la formazione
di legami idrogeno fra l'atomo di idrogeno del gruppo NH di un segmento
e l'ossigeno del gruppo carbonilico dell'altro segmento. Si ottiene in
questo modo la struttura riprodotta in basso in Figura 15. Nonostante l'effetto
stabilizzante dei legami idrogeno, tuttavia, la struttura completamente
planare mostrata non esiste nelle proteine naturali a causa delle repulsioni
steriche che essa comporta fra i gruppi R dei due segmenti di catena affacciati.
Nella realtà le catene si contraggono attraverso rotazioni più
o meno significative attorno ai legami singoli
N-C* e C*-C(carbonilico)
che permettono di allontanare i gruppi R ingombranti e allentare la repulsione.
A causa di queste rotazioni la struttura da planare diventa corrugata dando
luogo a quella che è nota come struttura ![]() a pieghe o configurazione
a pieghe o configurazione ![]() .
Questo
tipo di struttura secondaria è rappresentata schematicamente in
Figura 16. Come si vede la struttura tridimensionale della configurazione
.
Questo
tipo di struttura secondaria è rappresentata schematicamente in
Figura 16. Come si vede la struttura tridimensionale della configurazione
![]() può
essere assimilata nel suo complesso
a una sorta di porta a soffietto (o fisarmonica) nella quale, sugli spigoli
formati dai vari piani, si trovano posizionati i carboni tetraedrici
C*.
In realtà, questo tipo di struttura è in grado di accomodare
gruppi R non troppo voluminosi. Questo è quello che accade, per
esempio, nel caso della fibroina, la proteina della seta, nella
quale è presente il 48% di glicina (R=H) e il 38% di serina (R=CH2OH)
e alanina (R=CH3).
può
essere assimilata nel suo complesso
a una sorta di porta a soffietto (o fisarmonica) nella quale, sugli spigoli
formati dai vari piani, si trovano posizionati i carboni tetraedrici
C*.
In realtà, questo tipo di struttura è in grado di accomodare
gruppi R non troppo voluminosi. Questo è quello che accade, per
esempio, nel caso della fibroina, la proteina della seta, nella
quale è presente il 48% di glicina (R=H) e il 38% di serina (R=CH2OH)
e alanina (R=CH3).
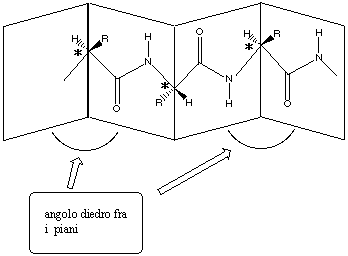
Figura 16. Struttura ![]() a pieghe o configurazione
a pieghe o configurazione ![]() di una catena proteica.
di una catena proteica.
Se i gruppi R diventano più ingombranti la struttura secondaria
preferita è quella indicata con il termine
![]() elica, certamente la struttura secondaria più importante per
le proteine naturali. In essa la catena polipeptidica è avvolta
a spirale a formare un'elica destrorsa con 3,6 amminoacidi per ogni giro
(il termine destrorsa sta a indicare che se si gira l'elica in senso
orario, questa si avvita allontanandosi dall'osservatore). Questa struttura,
riportata in figura 17, è stabilizzata da due fattori:
elica, certamente la struttura secondaria più importante per
le proteine naturali. In essa la catena polipeptidica è avvolta
a spirale a formare un'elica destrorsa con 3,6 amminoacidi per ogni giro
(il termine destrorsa sta a indicare che se si gira l'elica in senso
orario, questa si avvita allontanandosi dall'osservatore). Questa struttura,
riportata in figura 17, è stabilizzata da due fattori:
- Ogni gruppo ammidico della catena forma legami idrogeno con un gruppo ammidico sovrastante e uno sottostante lungo l'elica a una distanza di tre amminoacidi (per la precisione per ogni legame peptidico il gruppo N-H punta verso il basso approssimativamente parallelo all'asse dell'elica e il gruppo C=O in direzione opposta in modo da ottimizzare l'interazione O...H);
- i gruppi R ingombranti sono orientati verso l'esterno dell'elica per minimizzare le repulsioni.
Un esempio di ![]() -elica è
dato dalla
-elica è
dato dalla ![]() -cheratina
che costituisce la proteina dei peli, delle unghie e della lana. Un altro
esempio è la miosina che è la proteina dei muscoli.
-cheratina
che costituisce la proteina dei peli, delle unghie e della lana. Un altro
esempio è la miosina che è la proteina dei muscoli.
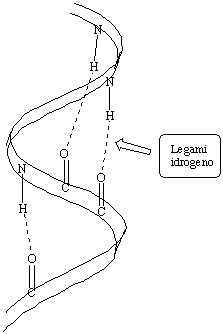
Figura 17. Struttura del tipo ![]() -elica.
Sono evidenziati i legami idrogeno fra i gruppi C=O e N-H.
-elica.
Sono evidenziati i legami idrogeno fra i gruppi C=O e N-H.
La struttura terziaria fa riferimento al ripiegamento nello spazio
dell'intera catena proteica. Anche se non esiste una separazione netta
fra struttura secondaria e struttura terziaria, si può dire che
la prima fa riferimento alla configurazione spaziale degli amminoacidi
vicini l'uno all'altro, mentre la seconda tiene conto della distribuzione
spaziale di tutti gli atomi di una catena. Così alcuni tratti della
catena possono essere presenti sotto forma di ![]() -elica
e altri come configurazione
-elica
e altri come configurazione ![]() , a seconda del
tipo di amminoacidi presenti, mentre i residui segmenti polipeptidici sono
caratterizzati da strutture non periodiche, diversamente dalle due precedenti,
chiamate conformazioni a spira o
ad ansa. L'intera catena
è poi avvolta in modo più o meno complesso a formare la struttura
terziaria.
, a seconda del
tipo di amminoacidi presenti, mentre i residui segmenti polipeptidici sono
caratterizzati da strutture non periodiche, diversamente dalle due precedenti,
chiamate conformazioni a spira o
ad ansa. L'intera catena
è poi avvolta in modo più o meno complesso a formare la struttura
terziaria.
Interazioni di varia natura giocano un ruolo importante nel determinare la struttura terziaria:
- I ripiegamenti della catena proteica avvengono in modo tale da orientare il maggior numero possibile di gruppi polari (gruppi idrofili) verso l'ambiente esterno acquoso e i gruppi non polari (gruppi idrofobi come quelli di valina, leucina, isoleucina, fenilalanina) verso le zone interne della proteina, il più lontano possibile dalle molecole d'acqua circostanti.
- Interazioni elettrostatiche fra siti positivi (come ione ammonio) e siti negativi (come ioni carbossilato).
- Legami idrogeno.
- Legami disolfuro.
Questi ultimi si formano fra i gruppi CH2SH di due molecole di cisteina. I gruppi SH si ossidano per formare un legame disolfuro -S-S- fra due segmenti avvolti della stessa catena o fra due catene, come accade, per esempio, nell'insulina umana. Questa proteina, in particolare, è costituita da due catene polipeptidiche: le catene A e B formate da 21 e 30 amminoacidi rispettivamente. Due legami -S-S- connettono le due catene in due diverse posizioni: un legame coinvolge la cisteina di A in posizione 7 e la cisteina di B nella stessa posizione, l'altro legame connette le due posizioni 20 e 19 di A e B rispettivamente. Inoltre un legame si instaura all'interno della catena A fra le posizioni 6 e 11. I legami sono indicati schematicamente in Figura 18.
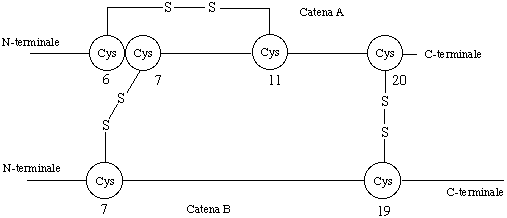
Figura 18. Rappresentazione schematica dell'insulina umana. Sono messi
in evidenza i ponti disolfuro intra-catena e inter-catene.
Alcune proteine, dette proteine globulari, sono avvolte in modo
molto complesso a formare una specie di gomitolo di forma approssimativamente
sferica. Un esempio di proteina globulare è la mioglobina,
una proteina preposta, insieme all'emoglobina, suo analogo
strutturale, al trasporto e all'immagazzinamento dell'ossigeno nei vertebrati
(l'emoglobina lega l'ossigeno molecolare nei polmoni e lo trasporta alla
mioglobina nei muscoli; quest'ultima immagazzina l'ossigeno fino a quando
esso non viene richiesto per le ossidazioni metaboliche). La struttura
tridimensionale della mioglobina è stata determinata in dettaglio.
Per questo lavoro fu assegnato il premio Nobel nel 1962 ai due ricercatori
britannici J.C. Kendrew e M.F.Perutz. Questa proteina è costituita
da una singola catena polipeptidica formata da 153 amminoacidi avvolta
su se stessa a formare una specie di scatola. Sono presenti lungo la catena
otto sezioni di ![]() -elica, separate da ripiegamenti
(anse) della catena, i quali includono circa il 75% del numero complessivo
di amminoacidi. Il ripiegamento della catena è tale da orientare
verso l'interno i gruppi idrofobi di fenilalanina, valina, isoleucina e
metionina, mentre la superficie esterna della molecola è rivestita
di catene idrofile come lisina, arginina e serina.
-elica, separate da ripiegamenti
(anse) della catena, i quali includono circa il 75% del numero complessivo
di amminoacidi. Il ripiegamento della catena è tale da orientare
verso l'interno i gruppi idrofobi di fenilalanina, valina, isoleucina e
metionina, mentre la superficie esterna della molecola è rivestita
di catene idrofile come lisina, arginina e serina.
Oltre ad appartenere alla classe delle proteine globulari, la mioglobina, come l'emoglobina, viene anche classificata come proteina coniugata. Le proteine coniugate includono una parte non proteica, che può essere, per esempio, uno zucchero, detto gruppo prostetico. Nel caso di mioglobina e emoglobina il gruppo prostetico è il gruppo eme che occupa una cavità della molecola. L'eme è costituito da uno ione Fe2+ che forma quattro legami di coordinazione con gli atomi di azoto di una molecola di porfirina (vedi Figura 19) e un quinto legame, approssimativamente perpendicolare al piano dell'anello porfirinico, con l'azoto dell'anello imidazolico di una istidina appartenente alla catena polipeptidica. Il sesto sito di coordinazione rimane così libero per coordinare una molecola di ossigeno, come mostrato a destra in Figura 19.
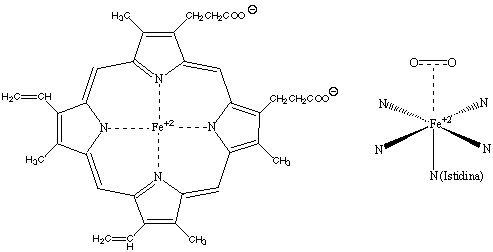
Figura 19. Anello porfirinico con al centro uno ione Fe+2
(a sinistra). Coordinazione complessiva dello ione Fe+2 nell'eme
(a destra).
Molte proteine di elevato peso molecolare sono in realtà degli aggregati di due o più catene proteiche e sono caratterizzate da una struttura quaternaria che corrisponde alla disposizione tridimensionale delle varie catene nell'aggregato. Un esempio di struttura quaternaria è dato nuovamente dall'emoglobina che è costituita da quattro catene separate: due di queste sono costituite da 141 amminoacidi e le rimanenti da 146 amminoacidi ciascuna. Le quattro catene, ognuna delle quali possiede un gruppo eme che può coordinare l'ossigeno, sono disposte in una struttura globale di tipo tetraedrico nella quale l'aggregazione delle quattro unità è stabilizzata principalmente da effetti idrofobici.